【祝贺】112lab在《Nature Communication》发表论文
化学与生物工程学院
生物信息学与调控网络本科生研究团队
杜宏武教授及其合作者 在《Nature Communication》上发表题为“OmicVerse: A Framework for Bridging and Deepening Insights Across Bulk and Single-Cell Sequencing”的文章。

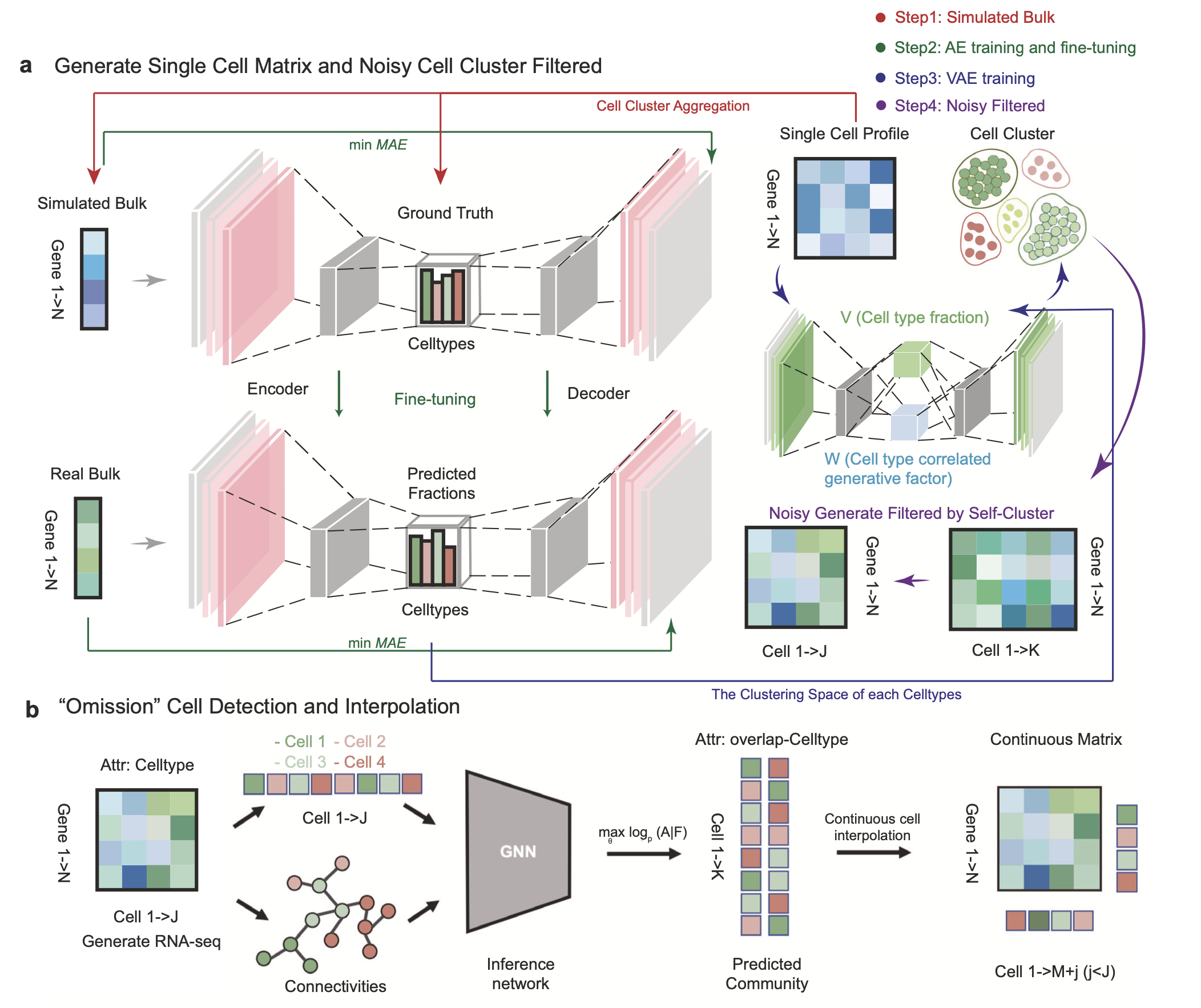
单细胞RNA测序(scRNA-seq)和Bulk RNA测序(RNA-seq)是探索细胞异质性、发育分化和疾病机制的重要技术。由于测序平台的技术限制以及酶解过程造成的细胞丢失,某些细胞类型在单细胞测序中有时会被遗漏,例如足细胞、中间祖细胞、神经元等。针对该问题目前已有一些解卷积算法提供了有益尝试,但仍然需要更多的算法研究以共同解决测序中的“遗漏”细胞问题。
近日,杜宏武教授团队提出了“BulkTrajBlend”算法,这是一种专门用于解决测序数据中“遗漏”细胞问题的新算法。BulkTrajBlend采用基于beta-变分自编码器和图神经网络的算法,从整体RNA测序中解卷单细胞数据,帮助在重建的单细胞景观中识别“遗漏”细胞,并用于增强单细胞的轨迹推断。
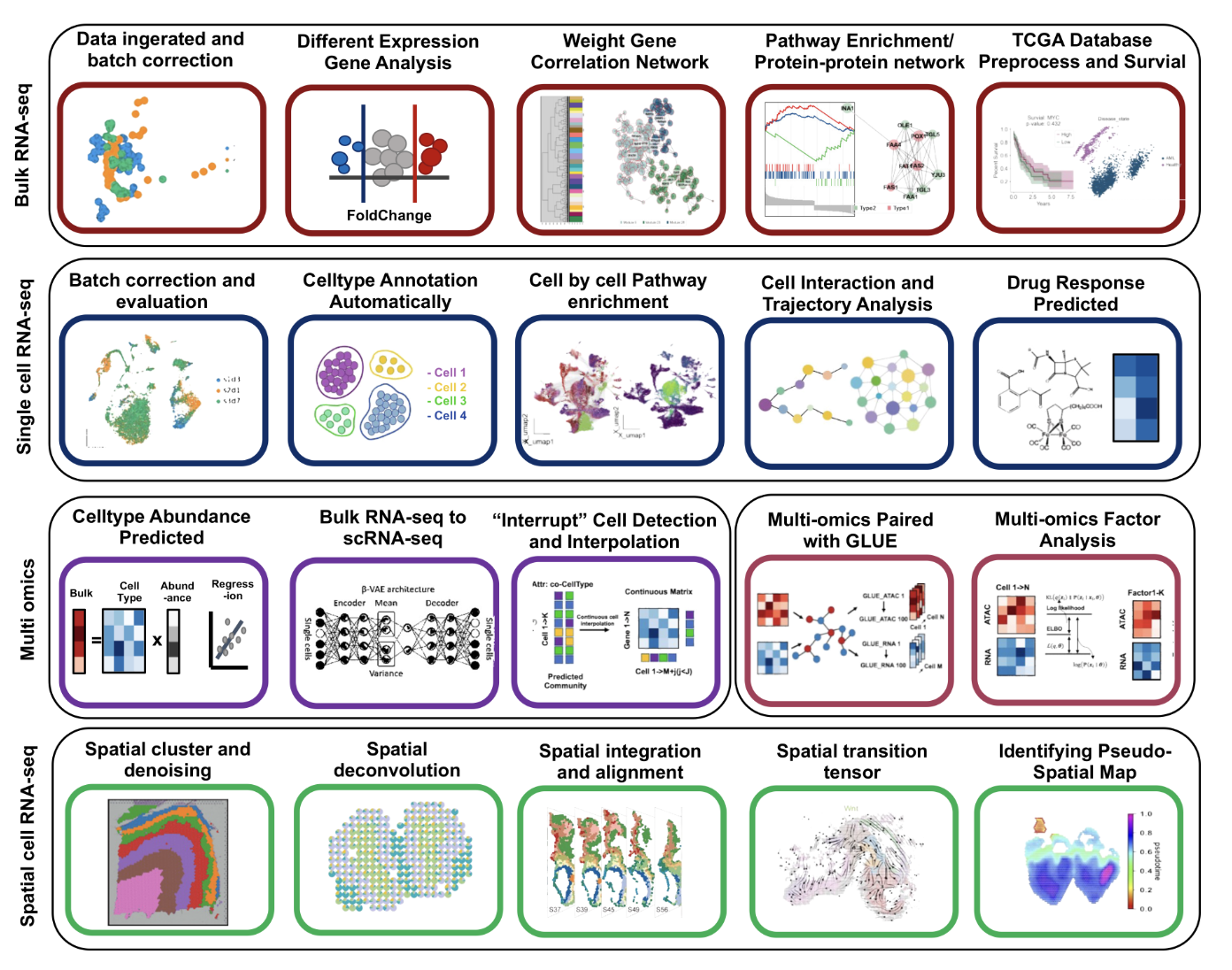
单细胞RNA测序常规的分析包括:差异表达分析、通路富集、基因共表达网络分析、细胞注释、细胞相互作用分析、细胞轨迹推断、基因集中的细胞状态评估以及单细胞RNA测序中的药物反应预测。但是,随着组学算法种类和数量的增加,选择合适且易用的工具反而变得越加困难,由于不同算法间缺乏统一的框架、数据间缺乏统一的标准化范式、不同算法的结果复现性较差,导致了数据分析和计算效率不尽如人意。
针对这一问题,杜宏武教授团队设计实现了“OmicVerse”框架,这是世界范围内首个基于Python完成Bulk RNA测序和单细胞RNA测序全分析任务的框架。该框架统一了不同算法的输入输出,统一了数据的标准化范式,并且规范了40+不同算法的分析流程,提供了一个用户友好的API接口。同时,该框架还提供了具有一定艺术表现力的可视化结果输出,为我国单细胞RNA测序的规范有序发展提供了新策略。
北京科技大学化学与生物工程学院2022级博士研究生曾泽华、清华大学生物医药与健康工程研究院2022级硕士研究生马雨晴、西湖大学生命科学学院2023级博士研究生胡磊为论文的共同第一作者(以上三名同学均在北京科技大学取得本科学位)。此外,除第一作者和通讯作者外,本文其他作者均为北京科技大学在校本科生。
该工作得到了科学技术部、北京科技大学本科生科研创新项目(SRTP)联合资助。
 杜宏武,北京科技大学化学与生物工程学院教授,博士生导师,大兴研究院执行院长,国家“科技奥运先进个人”,教育部“新世纪优秀人才”,中关村“新国门领军人才”,长期从事高通量芯片检测,干细胞治疗等方面的研究工作。
杜宏武,北京科技大学化学与生物工程学院教授,博士生导师,大兴研究院执行院长,国家“科技奥运先进个人”,教育部“新世纪优秀人才”,中关村“新国门领军人才”,长期从事高通量芯片检测,干细胞治疗等方面的研究工作。